单细胞转录组(scRNA-seq)典型实验流程
-
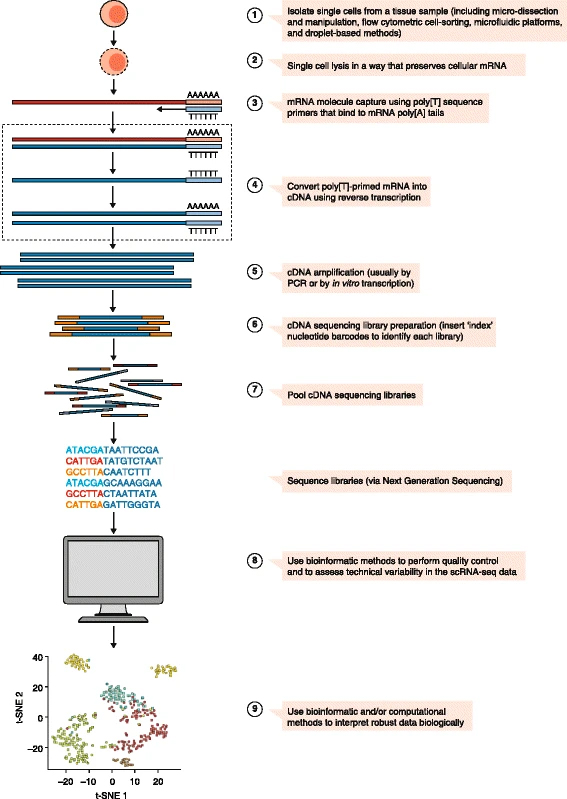
单细胞分离自组织样本获取单个细胞(显微切割、流式分选、微流控/液滴平台等)。
-
细胞裂解在保护细胞内 mRNA 的条件下进行裂解。
-
mRNA 捕获用 poly(T) 引物结合 mRNA 的 poly(A) 尾实现分子捕获。
-
反转录将 mRNA 逆转录为 cDNA(poly(T) 引物起始)。
-
cDNA 扩增PCR 或体外转录(IVT)放大低起始量的 cDNA。
-
文库构建与加索引制备测序文库并加入样本条形码(index)。
-
文库混合多样本文库混合上机,提升测序通量与性价比。
-
高通量测序采用 NGS 平台获取转录本序列读段。
-
生信分析质控、去噪、归一化与降维(如 t-SNE/UMAP),聚类与注释,差异分析与通路解析等。
随着高通量测序技术的进步,单细胞转录组分析(scRNA-seq)为研究者提供了在单细胞分辨率下测量基因表达的能力, 有助于揭示细胞群体中的异质性、发育轨迹以及功能状态变化。
与传统的群体测序相比,单细胞转录组能够捕捉稀有细胞类型,解析细胞间的差异与联系, 在肿瘤免疫、组织发育、疾病机制研究等领域展现出巨大潜力。
我们将系统梳理单细胞转录组的分析流程,从数据采集、质控、降维聚类到功能解读, 帮助科研人员高效、精准地解析单细胞层面的分子信息。
数据准备与质量控制
目标:去掉低质量/双细胞,获得稳定可靠的表达矩阵。
关键检查:
● 细胞基因数 ● UMI ● 线粒体/核糖体比例 ● 双细胞检测(Doublets)
常用方法:
FastQC、CellRanger / STARsolo、Scrublet / DoubletFinder、阈值过滤
产出:
高质量的细胞×基因表达矩阵(count matrix)、质控报告与过滤参数记录
降维、聚类与细胞类型注释
目标:获得可解释的细胞群及其功能/空间含义,并为后续分析提供稳定标签。
降维:
● PCA(线性主成分,输入后续步骤) ● UMAP/t-SNE(非线性可视化,呈现簇结构) ● 变量特征选择(HVGs)与尺度化(Scale)
聚类:
● 图谱聚类:Louvain / Leiden(基于KNN图) ● 分辨率调优(resolution grid search) ● 过/欠聚类检查与合并(marker一致性、轮廓系数)
细胞类型注释:
● Marker基因规则库(CD3D、MS4A1、LYZ、COL1A1 等) ● 参考库:SingleR、CellTypist、scHCL ● 专家修订与亚群拆分(子簇marker复核)
与空间数据衔接(可选):
● 以注释簇为来源,映射到空间数据(如 Tangram / Cell2location)
产出:
低维坐标(PCA/UMAP/t-SNE)、聚类标签(簇ID)、细胞类型注释表、关键marker清单与可视化图件。
差异表达与功能富集
目标:找出关键标志物和核心通路,支持分型与机制推断。
差异表达(DE):
● 统计方法:Wilcoxon、MAST、edgeR / DESeq2(伪 bulk) ● 分组策略:簇间、条件间、时间点比较 ● 输出指标:log2FC、p 值、调整后 p 值(FDR)
功能富集:
● 数据库:GO、KEGG、Reactome、WikiPathways ● 方法:GSEA、GSVA、ORA(Over Representation Analysis) ● 结果呈现:条形图、气泡图、网络图
产出:
差异基因列表(带统计指标)、通路富集结果表、可视化图件(火山图、气泡图、网络图)及对应脚本。
拟时序与谱系轨迹
目标:解析细胞状态随时间或分化过程的动态变化及分支关系。
方法:
● Monocle3(基于学习图构建拟时序) ● Slingshot(整合聚类与轨迹推断) ● PAGA(图抽象简化轨迹结构) ● Palantir(概率式轨迹与终末状态预测)
输出:
● 拟时序坐标与轨迹图 ● 分支节点与分化路径 ● 动态基因模块(随时间变化的表达模式)
应用:
发育与分化过程研究、耐药演化机制解析、免疫活化与衰竭路径分析。
细胞—细胞通讯
目标:解析配体-受体信号轴与网络结构,揭示细胞互作及微环境重塑机制。
方法:
● CellChat(全局通信概率与主导通路识别)
● CellPhoneDB(配体-受体配对统计与显著性检验)
● NicheNet(配体驱动的下游基因预测)
● LIANA(多工具整合评估,统一评分框架)
输出:
● 配体-受体网络与强度矩阵(群体/簇间通信图)
● 关键信号轴:如 TGF‑β、CXCL/CCR、EGFR、IFN 等
● 受体端下游靶基因与受影响通路(可视化:弦图、热图、气泡图)
应用:
肿瘤-免疫互作解析、免疫逃逸与炎症放大环识别、微环境重塑与治疗靶点筛选、疗效/耐药相关通信模式比较。
批次效应与多组学整合
目标:消除技术/样本来源差异,统一坐标系;融合多模态信息提升解析深度与稳定性。
批次校正:
● Harmony(嵌入对齐,速度快、稳健)
● MNN / FastMNN(互为最近邻匹配,减少批次偏移)
● BBKNN(图构建层面的批次平衡)
● Seurat Anchors / Scanorama(锚点配准,跨平台整合)
多组学整合:
● scRNA + ATAC:Seurat v4 WNN / ArchR(转录 × 染色质可及性共解析)
● 蛋白(CITE‑seq):ADT 与 RNA 表达联合建模(WNN/TotalVI)
● 甲基化/空间:Tangram、Cell2location(将单细胞注释映射到空间坐标)
● 代谢/配体‑受体:与代谢通路/通讯网络耦合,形成多层证据链
质量评估与可视化:
● 批次标签在 UMAP/TSNE 上的混合度(kBET、LISI 指标)
● 保真度:已知生物亚群的聚类纯度与marker一致性
产出:
统一整合后的低维嵌入与邻接图、跨批次聚类标签、多组学联合注释表与关联图件(WNN热图、共嵌入UMAP、对齐前后对比图)。
结果可视化与交付
目标:以直观、美观且可溯源的方式呈现分析成果,方便科研复现与成果产出。
常见图型:
● UMAP / t-SNE(细胞群分布)
● 火山图(差异基因显著性)
● 气泡图(marker表达模式)
● 热图(基因表达矩阵、聚类特征)
● 轨迹图(拟时序与分支结构)
● 通讯弦图 / 网络图(细胞间信号互作)
交付内容:
● 分析报告(PDF/HTML,含图文解读)
● 细胞类型注释表、聚类标签表
● 差异表达基因列表(含统计指标)
● 通路富集与功能注释结果表
● 关键图件源文件(SVG/PNG,高分辨率)
● 全流程复现脚本与参数记录(便于复现)
可选增强:
● 在线交互式浏览(Shiny App / CellxGene / Vitessce)
● 与团队知识库对接,便于后续复用
- 肿瘤研究:解析分型与耐药机制,预测免疫治疗应答
- 免疫学:追踪亚群谱系、活化或衰竭状态,评估疫苗免疫效果
- 组织发育与再生医学:揭示谱系决定与微环境调控机制
- 罕见病与自身免疫病:定位致病细胞群并解析分子特征
- 药效与毒理学:分析给药前后细胞状态变化,评估靶向性与安全性
单细胞转录组分析为科研人员提供了细胞层级的精准视角,帮助揭示生物过程中的关键机制与细胞多样性。 它不仅是基础研究的重要工具,也在临床转化、药物开发等领域展现出广阔的应用前景。
